ANÁLISIS BIOINFORMATICO DE LAS SECUENCIAS DE AMINOÁCIDOS DE DIVERSAS FOSFOLIPASAS D
DOI:
https://doi.org/10.33017/RevECIPeru2004.0004/Palabras clave:
Fosfolipasa D, Bioinformática, bases de datos biológicas, alineamiento múltiple.Resumen
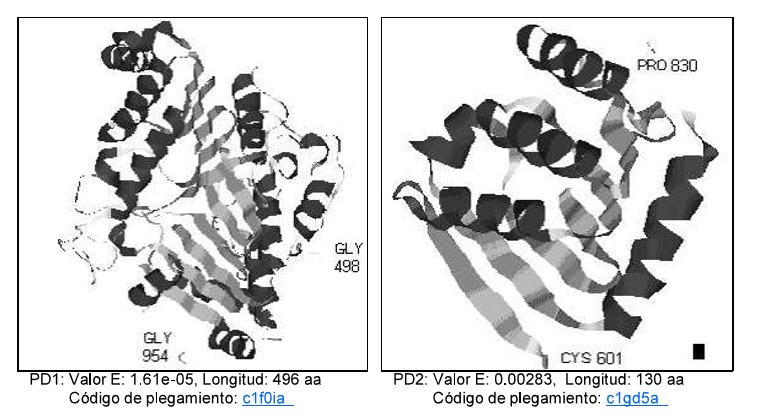
La Fosfolipasa D es una enzima presente en todos los organismos eucariotas, su papel en los mecanismos de señalización intracelular ha sido bastante estudiado en plantas, no obstante el rol que cumplen en mamíferos todavía no ha sido destacado respecto a otros moduladores de la señalización intracelular. Por ello se analizo insilico las secuencias de aminoácidos de las isoformas humanas D1 y D2 de la Fosfolipasa D, obteniendo la predicción de sus propiedades químicas responsables de sus diferentes niveles de acción. Usando el programa MACAW realizamos alineamientos múltiples locales de secuencias obtenidas de la base de datos NR del NCBI correspondientes a la “Fosfolipasa D” de varias especies encontrando sólo un segmento conservado entre las secuencias de origen bacteriano, y ocho entre las secuencias de origen vegetal. Se detecto además tres segmentos de secuencia de aminoácidos comunes en secuencias de especies de distante origen filogenético, la visualización de estos segmentos en la estructura 3D de la fosfolipasa D de la Bacteria Streptomyces Sp nos revelo que los residuos de aminoácidos altamente conservados de estos segmentos se localizan cercanamente formando una zona en la proteína que probablemente contiene los residuos esenciales para su actividad.